¿Qué es el ARN no codificante y para qué sirve?
Hace muchos años que sabemos que el ADN es la molécula que almacena la información genética de cualquier ser vivo, la que se transmite de generación en generación, y la que hace que los hijos se parezcan a sus padres. Cada célula de nuestro cuerpo, tan pequeña que sólo es visible bajo un microscopio, almacena nada más y nada menos que alrededor de 2 metros de ADN. Esta larga cadena está compuesta por 4 tipos de nucleótidos, que podemos imaginar como piezas de 4 colores diferentes.
En una célula viva, la información almacenada en el ADN se pone en práctica para producir sus moléculas funcionales, y esto ocurre siguiendo un código que viene determinado por la combinación de colores de las piezas que conforman el ADN. Ésta es la base del Dogma Central de la Biología Molecular, que fue descrito en los años 60. Desde entonces sabemos que la combinación de colores de un fragmento de ADN, lo que llamamos gen, se traslada primero a la producción de una molécula muy similar y de secuencia complementaria, llamada ARN, y que el código de colores en el ARN se traduce en la producción de los bloques que conforman las proteínas, las moléculas de la célula que permiten su funcionamiento [1].
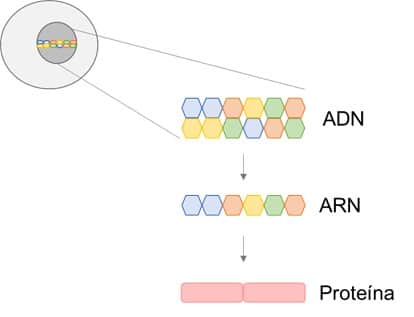
Años después, y con la evolución de la tecnología, la comunidad científica consiguió conocer la secuencia entera del ADN humano y de otros organismos. Por sorpresa, se vio que las secuencias de los genes clásicos o codificantes, que culminan con la producción de proteínas, representaban un porcentaje muy pequeño del ADN en toda su longitud [2], [3]. Conociendo las bases de la evolución, y la sabiduría de la naturaleza, ¿cómo podía ser que las células gastaran tanta energía en replicar y mantener una larguísima cadena de ADN, que en su mayoría no sirviera para nada? La sorpresa continuó cuando, con la secuenciación de las especies de ARN producidas en las células, se vio que la cantidad de ARN producido desde los genes codificantes era insignificante, pues el ADN estaba produciendo ARN también en las zonas que fueron consideradas como basura, o no codificantes [4], [5]. Si el ADN basura ya podía considerarse un gasto energético innecesario, ¿era posible también mantener la producción masiva de ARN basura?
El campo de los ARN no codificantes resultó revolucionario. Para entenderlo, los científicos hicieron una clasificación de estas especies de ARN encontradas, basada en su longitud, y empezaron a estudiar su funcionalidad [6]. Parece ser que las características bioquímicas del ARN confieren a esta molécula la capacidad de realizar funciones propias, y diferentes a las de las proteínas, aparte de su función clásica de retrasmitir la información que se encuentra en el ADN para la producción de proteínas. De hecho, el papel de los ARN no codificantes ha resultado ser esencial en diferentes procesos celulares. Pero es que, además, estas funciones alternativas son muy diversas, respondiendo a la gran diversidad de ARN no codificantes que se producen. Por ejemplo, los microARNs, de unos 20 nucleótidos de longitud, son capaces de reconocer especies concretas de ARNs codificantes, para inducir su degradación y así controlar su traducción a proteína, que puede ser necesaria o no dependiendo de las condiciones de la célula en ese determinado momento [7]. En cambio, las funciones de los que llamamos ARN no codificantes largos (lncARN), de más de 200 nucleótidos de longitud, suelen residir en el núcleo de las células, para regular la funcionalidad del ADN, que está acompañado de proteínas y otras especies de ARN [8]. Un ejemplo clásico de lncARN es Xist (X-inactive-specific transcript), que tiene la función de reprimir la actividad de uno de los dos cromosomas X que tienen las mujeres, para evitar la puesta en práctica de información redundante, que en cambio no se encuentra en los hombres, que tienen en cada célula un cromosoma X y uno Y [9]. Por su estructura podría parecer un ARN codificante, pero sabemos que Xist, al igual que cualquier ARN no codificante, no se traduce a proteína, porque su código de colores no tiene sentido para la maquinaria de producción proteica.
A día de hoy se siguen descubriendo nuevas funcionalidades de diferentes especies de ARN, y su estudio detallado ha llevado a entender su versatilidad como moléculas reguladoras. Mientras que la producción de proteínas implica varios pasos de señalización, la producción y degradación de ARN es muy rápida, lo que le permite a la célula responder a estímulos de manera mucho más eficaz. De hecho, no solamente los ARN no codificantes tienen esta propiedad, sino que cualquier ARN que se esté produciendo, incluso los clásicos ARN codificantes o mensajeros (mARN), tiene la capacidad de actuar como molécula reguladora, por ejemplo modulando la actividad del ADN u otros procesos celulares, a través de mecanismos que todavía no terminamos de entender [10], [11].

Comprendemos ahora que el campo del ARN está todavía en expansión, y es por eso por lo que algunos curiosos siguen invirtiendo recursos en esta investigación aparentemente no aplicada en la medicina, pues de momento no parece que vaya a dejar de sorprendernos.
[1] F. H. Crick, “On protein synthesis.,” Symp. Soc. Exp. Biol., vol. 12, pp. 138–63, 1958.
[2] E. S. Lander et al., “Initial sequencing and analysis of the human genome,” Nature, vol. 409, no. 6822, 2001.
[3] J. Craig Venter et al., “The sequence of the human genome,” Science (80-. )., vol. 291, no. 5507, 2001.
[4] P. Carninci et al., “The Transcriptional Landscape of the Mammalian Genome,” Science (80-. )., vol. 309, no. 5740, pp. 1559–1563, Sep. 2005.
[5] S. Djebali et al., “Landscape of transcription in human cells,” Nature, vol. 489, no. 7414, pp. 101–108, Sep. 2012.
[6] S. Hombach and M. Kretz, “Non-coding RNAs: Classification, biology and functioning,” Adv. Exp. Med. Biol., vol. 937, pp. 3–17, 2016.
[7] A. Kozomara, M. Birgaoanu, and S. Griffiths-Jones, “MiRBase: From microRNA sequences to function,” Nucleic Acids Res., vol. 47, no. D1, 2019.
[8] F. P. Marchese, I. Raimondi, and M. Huarte, “The multidimensional mechanisms of long noncoding RNA function,” Genome Biol., vol. 18, no. 1, p. 206, Oct. 2017.
[9] C. J. Brown et al., “The human XIST gene: analysis of a 17 kb inactive X-specific RNA that contains conserved repeats and is highly localized within the nucleus.,” Cell, vol. 71, no. 3, pp. 527–42, Oct. 1992.
[10] L. Skalska, M. Beltran-Nebot, J. Ule, and R. G. Jenner, “Regulatory feedback from nascent RNA to chromatin and transcription,” Nat. Rev. Mol. Cell Biol., vol. 18, no. 5, pp. 331–337, May 2017.
[11] L. Statello, C. J. Guo, L. L. Chen, and M. Huarte, “Gene regulation by long non-coding RNAs and its biological functions,” Nat. Rev. Mol. Cell Biol., vol. 22, no. 2, pp. 96–118, Feb. 2021.







