El ARN naciente como regulador de la replicación del genoma humano
En Julio de 2023 se ha publicado, en la revista Nature Communications, un artículo de biología molecular en el que demostramos que las moléculas de ARN en producción, en el núcleo de las células, tienen un papel regulador sobre la replicación del ADN, un proceso celular esencial que lleva décadas siendo meticulosamente estudiado [1]. Vamos a ver por qué este descubrimiento es importante, y qué impacto tiene en la investigación actual.
La replicación del genoma humano
El genoma humano es el conjunto completo de instrucciones de ADN que nos identifica como especie. Para que un ser humano se desarrolle, y se mantenga como organismo multicelular durante su vida, las células que componen su cuerpo copian el ADN genómico que se halla en su núcleo, antes de dividirse en dos células idénticas. La replicación del ADN es entonces un proceso celular esencial, y como tal ha sido objeto de estudio desde que fue descubierto. Más aún, sabemos que una de las características que hacen que una célula sea cancerosa es su replicación descontrolada, retroalimentada por los errores en el ADN que deja como herencia, y culminando con la formación de tumores que pueden llegar a extenderse a otros tejidos [2], [3]. Así, entender la regulación de la replicación del ADN es esencial para diseñar nuevas terapias contra el cáncer.
El primer paso necesario para que el genoma humano se duplique es que la doble hebra de ADN se abra en determinados puntos de la cadena, para que la maquinaria de replicación pueda empezar a copiar la secuencia de nucleótidos que la conforman. Parece algo sencillo, pero la realidad es que los múltiples orígenes de replicación, los sitios específicos donde se inicia la replicación del ADN cada vez que una célula se divide, tienen que estar estrictamente regulados y coordinados entre sí, para conseguir la replicación completa del genoma, sin que falten trozos de ADN por replicar ni se repitan fragmentos ya copiados [4]. Además, la célula tiene que ser capaz de coordinar este proceso con otros procesos que están ocurriendo a la vez, pues el ADN, además de replicarse, se está transcribiendo, es decir, está generando ARN codificante y no codificante, está plegado y en contacto con otras regiones del ADN, e interactuando con gran diversidad de proteínas y moléculas de ARN, conformando una estructura dinámica conocida como cromatina, todo ello con impacto sobre su funcionalidad.
No es de sorprender entonces que la regulación de los orígenes de replicación sea muy compleja y, de hecho, lejos de ser entendida; una complejidad en realidad necesaria para que la célula pueda, de manera flexible, realizar correctamente todas sus funciones, adaptadas a cada situación fisiológica.
¿Cómo se regulan los orígenes de replicación?
La flexibilidad en la regulación de los orígenes de replicación es clara cuando los miramos de cerca en una población celular, a través de técnicas específicas que son capaces de detectarlos en base a sus características bioquímicas. Por un lado, los orígenes de replicación más frecuentemente activados en la población celular, se sitúan en zonas de transcripción activa [5]. Sin embargo, su activación no ocurre en las mismas posiciones genómicas de forma estricta. ¿Qué determina entonces la posición de los orígenes de replicación? ¿cómo la célula decide cuáles de los orígenes hacer funcionar, para conseguir la replicación completa y sin errores del genoma? Sabemos que la posición de los orígenes de replicación viene determinada por la unión, a esas zonas, del complejo de proteínas ORC (Origin Recognition Complex) [6]. Sin embargo, los conocimientos sobre su regulación son vagos, y de gran interés para la comunidad científica.
Lo que sabemos es que la regulación de los orígenes de replicación reside en la cromatina, esa compleja estructura compactada en el núcleo celular, compuesta por el ADN genómico, enrollado sobre unas proteínas llamadas histonas, y envuelto de otras especies de proteínas y de ARN. Mientras que determinadas modificaciones bioquímicas de las histonas, así como el mismo estado de compactación de la cromatina, favorecen que una determinada región del genoma se defina como un origen de replicación, ninguno de estos factores se encuentra representado en la mayoría de orígenes, de manera que tiene que haber algo más influyendo en esta regulación [6].
¿Qué han descubierto en este estudio?
En este estudio, hemos descubierto que, en las zonas del genoma donde está ocurriendo la transcripción, o la producción de ARN, la propia molécula de ARN está regulando la activación del origen de replicación, a través de su unión al complejo ORC.
Se revela entonces un novedoso mecanismo regulador de los orígenes de replicación, cuyo funcionamiento lleva estudiándose extensivamente desde hace más de 30 años. Las características bioquímicas del ARN, que le permiten a esta molécula tener funciones reguladoras, son en este caso importantes para controlar en qué zonas del genoma se inicia la replicación del ADN, de una manera dinámica y adaptada al estado transcripcional de la célula en ese determinado momento, lo cual es esencial para mantener la integridad y fidelidad genómica.
¿Cómo han llegado a estas conclusiones? ¿Cómo han estudiado el papel del ARN en la regulación de los orígenes de replicación?
En primer lugar, los autores hemos demostrado que la proteína ORC1, parte del complejo ORC, y la primera en asociarse al ADN para definir las posiciones de los orígenes de replicación [7], es capaz de unir directamente moléculas de ARN. La habilidad de ORC1 de interactuar con ARN es evidente in vitro, es decir, cuando mezclamos estos dos componentes en un tubo, pues se observa la formación de complejos proteína-ARN [8]. Además de esto, demuestramos que esta interacción está de hecho ocurriendo en las células humanas vivas. Por un lado, las moléculas de ARN en producción fueron marcadas con fluorescencia, y se vio, con microscopía de alta resolución, que colocalizan en el espacio junto a ORC1, que también fue marcada con fluorescencia. Por otro lado, a través de dos técnicas complementarias y de gran resolución, secuenciamos e identificamos qué especies concretas de ARN se asocian directamente a ORC1 en la célula. Son especies de ARN codificante, de una longitud mayor a la media, y abundantes, con altos ritmos de transcripción o producción.
Una vez establecido que ORC1 interactúa con ARN en las células vivas, demostramos que esta interacción es necesaria para que haya una óptima activación de los orígenes de replicación. Por un lado, los ARNs que interactúan con ORC1 de manera normal, fueron específicamente degradados en las células, y la actividad de los orígenes de replicación fue medida en esas condiciones. A través de diferentes técnicas moleculares, capaces de medir de diferentes formas la actividad de los orígenes de replicación, se vio que la ausencia de las especies de ARN que normalmente interactúan con ORC1 tiene un efecto negativo sobre la iniciación de la replicación del ADN. En estas condiciones, los orígenes de replicación más frecuentes, situados en zonas de transcripción activa, bajan su actividad. Concretamente, esto ocurre preferentemente en las zonas del ADN que están produciendo los ARNs experimentalmente desregulados. Estos datos sugieren que el ARN en transcripción, y cercano al ADN, está regulando, en su zona de producción, la formación de un origen de replicación a través de su unión con ORC1.
Además demostramos la importancia de la unión de ORC1 con ARN para la activación normal de orígenes, añadiendo a las células una forma mutante de ORC1 que es incapaz de unirse a moléculas de ARN. Simplemente sustituyendo artificialmente 3 de los bloques, o aminoácidos, que componen ORC1, se produce una forma de ORC1 que pierde la capacidad de unirse a ARN, lo cual es evidente in vitro, y también in vivo, o en las células vivas. Por ejemplo, detectamos que esta forma mutante de ORC1 pierde la colocalización con ARN naciente en experimentos de microscopía de alta resolución. Además, siguiendo un protocolo paralelo con la proteína ORC1 normal, no se pudo purificar ARN unido directamente a esta forma mutante de la proteína. Al igual que en los experimentos de degradación de los ARN normalmente unidos a ORC1, el uso de la forma mutante de ORC1, sin unión a ARN, resulta en una activación deficiente de los orígenes de replicación, afectado principalmente a los que se encuentran en los genes que producen los ARN más abundantes, y unidos a ORC1 en condiciones normales.
El estudio culmina con la explicación mecanística de esta novedosa regulación de los orígenes de replicación. Tanto con el uso del mutante de unión a ARN, como con la degradación específica de especies de ARN unidas a ORC1, logramos relacionar el defecto en la activación de los orígenes con una alteración en la disociación de ORC1 de la cromatina. En el artículo se demuestra que el ARN regula la fosforilación de ORC1, una modificación bioquímica que es necesaria para modular la interacción de ORC1 con el ADN, y que por lo tanto afecta su función correcta en la regulación de los orígenes de replicación.
En resumen, el artículo propone que el ARN en proceso de producción interactúa directamente con ORC1, favoreciendo su disociación de la cromatina, necesaria para la activación del origen de replicación que se encuentra en la región genómica que produce el ARN (ver figura). Entre células, la producción de ARN en cada situación es parecida, pero no idéntica, adaptada a sus particulares condiciones fisiológicas. Entonces, este nuevo mecanismo ayuda a explicar la variabilidad, de célula a célula, en la posición concreta de los orígenes de replicación a lo largo del genoma, clarificando también cómo las células pueden ser capaces de coordinar su patrón de transcripción con la replicación de su ADN, evitando conflictos entre ambos procesos y asegurando una duplicación completa de su material genético.
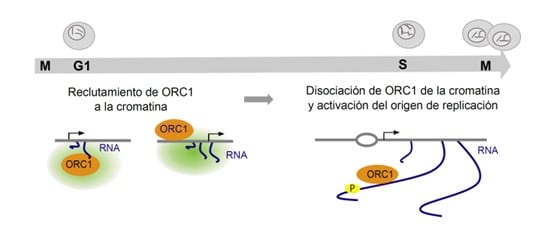
Figura modelo representando la unión de ORC1 a moléculas de ARN en producción, a lo largo del ciclo celular (Fases M, G1 y S). En el momento del ciclo donde la hebra de ADN se abre para iniciar su replicación (S), las moléculas de ARN interactuando con ORC1 regulan su fosforilación y disociación de la cromatina, favoreciendo la activación del origen de replicación en esa posición genómica
Perspectivas de futuro
Aunque este estudio representa un gran avance en la comprensión de la regulación de los orígenes de replicación del genoma humano, su entendimiento completo es todavía una meta lejana, que probablemente implica la combinación de otras funcionalidades de la cromatina y de otras proteínas del complejo replicativo que todavía desconocemos. Además, se expande el potencial regulador del ARN naciente como molécula no codificante. Este tipo de estudios nos abren los ojos ante la importancia de la investigación básica, pues entender la biología de nuestras células es el primer paso para pensar en nuevas terapias efectivas y personalizadas.
Referencias
[1] A. M. Mas et al., “ORC1 binds to cis-transcribed RNAs for efficient activation of replication origins,” Nat. Commun. 2023 141, vol. 14, no. 1, pp. 1–19, Jul. 2023.
[2] S. A. Hills and J. F. X. Diffley, “DNA replication and oncogene-induced replicative stress.,” Curr. Biol., vol. 24, no. 10, pp. R435-44, May 2014.
[3] S. Negrini, V. G. Gorgoulis, and T. D. Halazonetis, “Genomic instability — an evolving hallmark of cancer,” Nat. Rev. Mol. Cell Biol. 2010 113, vol. 11, no. 3, pp. 220–228, Mar. 2010.
[4] M. O’Donnell, L. Langston, and B. Stillman, “Principles and concepts of DNA replication in bacteria, archaea, and eukarya,” Cold Spring Harb. Perspect. Biol., vol. 5, no. 7, pp. 1–13, Jul. 2013.
[5] J. Sequeira-Mendes, R. Díaz-Uriarte, A. Apedaile, D. Huntley, N. Brockdorff, and M. Gómez, “Transcription Initiation Activity Sets Replication Origin Efficiency in Mammalian Cells,” PLoS Genet., vol. 5, no. 4, p. e1000446, Apr. 2009.
[6] M. Fragkos, O. Ganier, P. Coulombe, and M. Méchali, “DNA replication origin activation in space and time,” Nat. Rev. Mol. Cell Biol., vol. 16, no. 6, pp. 360–374, May 2015.
[7] J. Méndez, X. H. Zou-Yang, S.-Y. Kim, M. Hidaka, W. P. Tansey, and B. Stillman, “Human Origin Recognition Complex Large Subunit Is Degraded by Ubiquitin-Mediated Proteolysis after Initiation of DNA Replication,” Mol. Cell, vol. 9, no. 3, pp. 481–491, Mar. 2002.
[8] S. Hoshina et al., “Human origin recognition complex binds preferentially to G-quadruplex-preferable RNA and single-stranded DNA,” J. Biol. Chem., vol. 288, no. 42, pp. 30161–30171, 2013.
About the Author: Aina M. Mas
One Comment
Leave A Comment Cancelar la respuesta
Este sitio usa Akismet para reducir el spam. Aprende cómo se procesan los datos de tus comentarios.








El ARN naciente, también conocido como ARN primario (pre-ARNm), desempeña un papel importante en la regulación de la replicación del genoma humano. Aunque principalmente se asocia con la síntesis de proteínas, el proceso de transcripción del ADN a ARN naciente tiene implicaciones regulatorias clave en la replicación del genoma.
Él ARN naciente actúa como un intermediario en la expresión génica y puede influir en la replicación del genoma de diversas maneras: Splicing alternativo: Durante la transcripción, el ARN naciente puede someterse a splicing alternativo, donde se eliminan o se combinan diferentes exones.