¿Quién decide qué genes se expresan y cuáles no en la célula?
Soy Miriam Recalde y hoy en Dciencia voy a hablaros de un estudio que hemos publicado este verano en la revista Nucleic Acids Research (NAR). Este trabajo se ha llevado a cabo en el Programa de Hepatología del Centro de Investigación Médica Aplicada de la Universidad de Navarra y ha sido dirigido por la Dra. Carmen Berasain y la Dra. María Arechederra.
¿Qué hemos descubierto?
Se trata de un estudio de biología molecular y celular donde describimos una nueva e inesperada función para la proteína SLU7. En trabajos anteriores ya habíamos descrito que SLU7 regula la expresión génica a distintos niveles (transcripción y splicing). En este nuevo estudio describimos que SLU7 es esencial para mantener la regulación génica ¡a nivel epigenético! Integrando todo podemos decir que SLU7 tiene un papel fundamental en decidir qué genes se expresan y cuales no en una célula.
Pero… ¿qué significa todo esto? Y ¿qué implica? Voy a intentar explicároslo poco a poco.
Regulación de la expresión génica: del ADN a las proteínas
En nuestro cuerpo tenemos millones de células, todas ellas con la misma información en el ADN, sin embargo, cada célula es y actúa de manera diferente, por ejemplo, no es lo mismo una célula de la piel que una neurona (célula del cerebro), por la misma razón que una célula cancerosa actúa de manera diferente a una célula normal. Y esto se debe a que en cada célula se leen o expresan genes diferentes dando lugar a células con características y funciones específicas.
Regulación de la expresión génica. Es el proceso encargado de determinar qué genes se expresan en cada célula. Mediante el proceso denominado transcripción, la información contenida en fragmentos de ADN (llamados genes) se transfiere a una molécula de ARN, dando lugar a moléculas de ARN mensajero (ARN-m) que mediante el proceso de la traducción se convierten en proteína o puede dar lugar a moléculas de ARN no codificantes (ARN-nc), es decir que no llegan a formar proteína. De hecho, la mayor parte del genoma humano tras su procesamiento corresponde a estas últimas moléculas de ARN-nc.
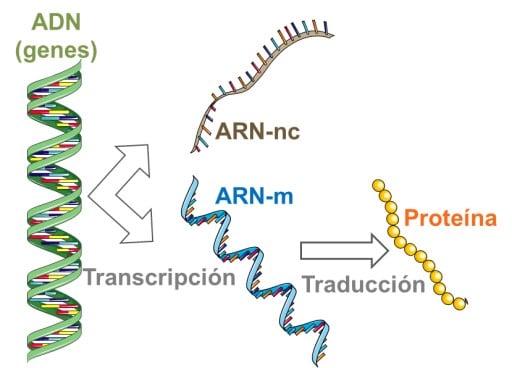
¿Cómo es esta “regulación de la expresión génica”? La regulación de la expresión génica es un proceso muy muy complejo y está controlado por diferentes proteínas que actúan a distintos niveles:
- Factores epigenéticos. Proteínas que actúan poniendo y quitando una serie de marcas químicas en el ADN sin modificar su secuencia. Dependiendo de la presencia o ausencia de las marcas y del tipo de marca, dejan el ADN “abierto” para que se pueda leer y traducir a proteínas o por el contrario el ADN queda “cerrado” y la información de ese fragmento de ADN no se va a expresar.
- Factores de transcripción. Una vez un fragmento de ADN ha quedado abierto, las proteínas que actúan como factores de transcripción se unen a dicho fragmento para impulsar la transcripción y generar así el ARN-m, o mantenerse unidos reprimiendo la expresión del gen.
- Factores de splicing: Una vez generada la molécula de ARN-m se realizan una serie de modificaciones en ella para hacerla estable y que llegue a traducirse a proteína, o por el contrario se realizan modificaciones que hacen que se degrade la molécula de ARN-m.
- Modificadores postraduccionales. Una vez tenemos la proteína, ésta también puede sufrir diferentes modificaciones que le marcan el futuro. Estas modificaciones determinan su función, localización dentro de la célula o su vida media.

¿Y qué relevancia tiene? La regulación de este proceso es muy importante ya que va a determinar qué proteínas se expresan en cada célula. Y es la presencia y función de esas proteínas las que van a dar forma a la célula y determinan su función, si algo no funciona bien, la célula deja de ser ella, pierde su identidad, se “desdiferencia o puede morir. Mientras que en las células normales diferentes sistemas de control aseguran la expresión génica para que los genes apropiados se expresen en los momentos adecuados, en distintas enfermedades y en cáncer este procedo está desregulado, de manera que se expresan genes que deberían estar silentes y se inhiben genes que deberían expresarse, haciendo que la célula no realice de forma correcta sus funciones o por ejemplo en el caso del cáncer sea capaz de crecer de forma descontrolada. Un ejemplo de esta desregulación en las células tumorales serían las alteraciones epigenéticas, en concreto alteraciones en las marcas de metilación del ADN. Encontramos una hipometilación global del ADN (se colocan menos grupos metilos unidos al ADN) que provoca que el ADN quede “abierto” y se expresan genes que favorecen la transformación maligna de la célula llamados oncogenes. Por el contrario, las marcas de metilación del ADN se acumulan en algunas regiones, cerrando el genoma, e inhibiendo la expresión de genes que actúan normalmente como genes supresores de tumores.
¿Quién es SLU7 y cómo regula la expresión génica?
Desde hace años, en nuestro grupo de investigación trabajamos caracterizando las funciones de la proteína SLU7. Hemos descrito que SLU7:
- Es necesario para mantener la expresión y función de factores de transcripción esenciales para preservar y mantener diferenciados a los hepatocitos (células principales del hígado) asegurando la correcta función del hígado adulto (Elizalde et al. 2014) y protegiéndolo frente al daño (Gárate-Rascón et al. 2021).
- Es esencial para la supervivencia de las células tumorales (Urtasun et al. 2016), de forma que, inhibir su expresión induce la muerte de diferentes tipos de células cancerosas.
- Es una proteína necesaria para prevenir la inestabilidad genómica y asegurar la correcta división de la célula (Jiménez et al. 2019).
¿Cómo lleva a cabo SLU7 todas estas funciones? Regulando la expresión génica. Hasta ahora habíamos descrito que SLU7 actuaba a dos niveles regulando la transcripción y el splicing.
Pero en este último trabajo demostramos que SLU7 también regula la expresión génica a nivel epigenético. Como hemos dicho, la epigenética son marcas químicas que se ponen o se quitan en el ADN y que determinan la expresión de los genes sin modificar la secuencia de ADN. Estas marcas epigenéticas son llevadas a cabo por distintas proteínas o complejos de proteínas. Un ejemplo de marca son los “grupos metilo” (-CH3). Cuando el ADN se metila, el fragmento donde ha tenido lugar la marca queda compacto y no se expresará el gen, por el contrario cuando no está presente la marca de metilación, se abre el ADN dando lugar a la expresión del gen. ¿Y qué tiene que ver SLU7 con esta metilación de ADN? Lo que hemos descubierto es que SLU7 forma parte del grupo de proteínas encargado de llevar a cabo la metilación del ADN. Cuando disminuimos o eliminamos los niveles de SLU7, este complejo se desarma y se degradan las proteínas que forman parte del mismo, produciéndose una metilación aberrante en el ADN y por tanto una expresión de genes anormal en la célula. Hemos demostrado que esta regulación de la metilación del ADN por SLU7 es importante cuando la célula se está dividiendo, es decir, SLU7 es muy importante por ejemplo para las células tumorales. En este trabajo hemos utilizado células tumorales de muy diversos orígenes (hígado, colón, mama, pulmón, glioblastoma, etc) observando que en todas ellas SLU7 controla la metilación del ADN.

En resumen
Nuestro trabajo en conjunto define a SLU7 como un factor pleiotrópico, es decir, que regula múltiples funciones no relacionadas, representando una conexión entre los distintos niveles de regulación de la expresión génica, desde la remodelación epigenética del ADN a la modulación de la transcripción y del splicing del mARN. Nuestros datos muestran que SLU7 es uno de los “jefes” de la célula, necesario para controlar a muy diferentes niveles qué genes se expresan y cuales no, siendo fundamental para mantener la identidad celular y para la supervivencia de las células tumorales.
Podéis encontrar el artículo original en:
https://academic.oup.com/nar/article/49/15/8592/6332818
Recalde M, Gárate-Rascón M, Elizalde M, Azkona M, Latasa MU, Bárcena-Varela M, Sangro B, Fernández-Barrena MG, Ávila MA, Arechederra M, Berasain C. The splicing regulator SLU7 is required to preserve DNMT1 protein stability and DNA methylation. Nucleic Acids Res. 2021 Sep 7;49(15):8592-8609. doi: 10.1093/nar/gkab649. PMID: 34331453; PMCID: PMC8421144.