CRISPR-Cas systems como nuevos antimicrobianos
CRISPR es el acrónimo del inglés “Clustered Regularly Interspaced Short Palindromic Repeats”, que se puede traducir por algo así como “repeticiones cortas palindrómicas regularmente interespaciadas y agrupadas” (en una misma región del genoma). Junto con CRISPR aparecen “CRISPR-associated (Cas) proteins” que son las proteínas asociadas a este sistema para que sea funcional.
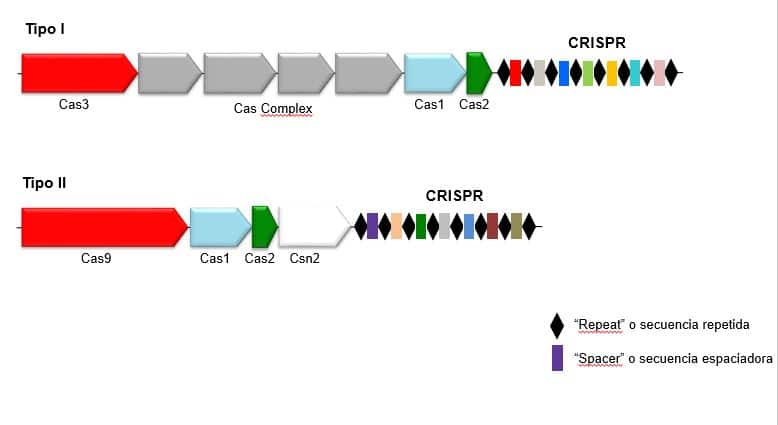
Representación esquemática del locus de los sistemas CRISPR-Cas Tipo I y Tipo II. En rojo el gen que da identidad a cada sistema: Cas3 para el Tipo I y Cas9 para el Tipo II. En azul y verde los genes universales Cas1 y Cas2. Los rombos negros representan las secuencias repetidas o “repeats” y los cuadrados representan las secuencias espaciadoras o “spacers”, cada color representando una distinta.
Los sistemas CRISPR representan el sistema inmune de las bacterias el cual actúa reconociendo de manera específica el material genético de los virus que las intentan infectar, atacándolo mediante cortes en su ADN y destruyéndolo. Sin embargo lo que ha hecho famoso a CRISPR es su aplicación como herramienta de edición genómica, generalmente basada en la nucleasa Cas9.
La tecnología basada en CRISPR ha tenido un gran impacto en el ámbito científico durante los últimos 10 años, pero ha sido especialmente revolucionaria durante los últimos 5 dada su infinita aplicación para la edición genómica en todo tipo de organismos y campos de estudio, desde la mejora de fermentos lácteos de uso industrial hasta su aplicación en la salud humana. La base de esta tecnología radica en la capacidad de reprogramar los sistemas CRISPR, guiando a la nucleasa (enzima que corta el ADN), para que esta actúe específicamente en la región de ADN (ó ARN) deseada. Esta capacidad de reprogramar el sistema junto con su increíble especificidad lo ha convertido en toda una revolución para la edición genómica, y uno de los mayores descubrimientos científicos de los últimos años.
De forma nativa, CRISPR actúa como sistema inmune de las bacterias, donde la secuencia espaciadora o “spacer”, formada por 20-30 nucleótidos, indica a la nucleasa a que región del ADN invasor se debe unir para posteriormente cortarlo. Esta secuencia espaciadora va unida a una secuencia repetida o “repeat” que junto con otros componentes son los que físicamente interactúan con la nucleasa para guiarla. Estos componentes dependen del tipo de CRISPR, de los que actualmente hay descritas 2 clases y 6 subtipos, sin embargo el Tipo I y Tipo II son los más estudiados hasta la fecha
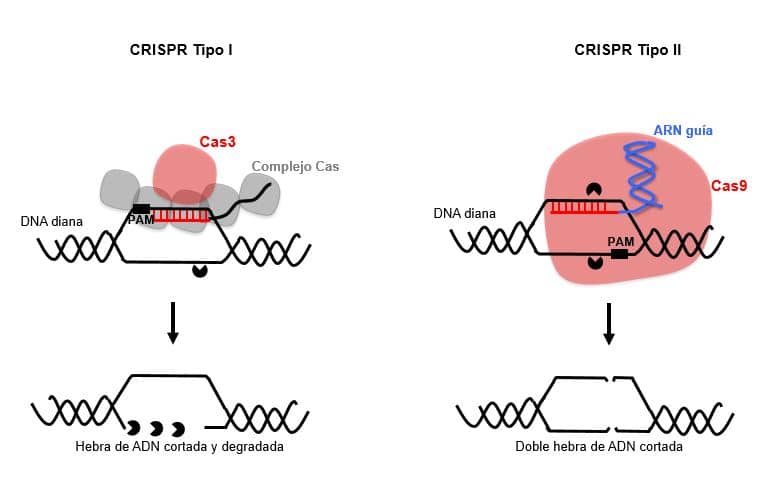
Representación esquemática del funcionamiento de los sistemas CRISPR-Cas Tipo I y Tipo II en su interacción con el ADN diana para cortarlo.
El sistema CRISPR Tipo II, cuyo signo de identidad es la endonucleasa Cas9, es el más estudiado y empleado universalmente, a la vez que el más sencillo, donde un par de componentes representan el ARN guía o “guide RNA” que dirige a la nucleasa. La capacidad de reprogramar este ARN guía se basa en mantener intacta toda su estructura y modificar sólo los 20-30 nucleótidos que forman la secuencia espaciadora, modificando así el objetivo diana de la nucleasa. La capacidad para reprogramar el sistema CRISPR fue desarrollada por Jennifer Doudna y Emmanuelle Charpentier y es lo que ha dado lugar a todo un abanico de opciones de potenciales aplicaciones en edición genética, desde bacterias hasta humanos pasando por todo tipo de organismos y en todos los campos de aplicación imaginables.
La especificidad de CRISPR radica en la perfecta homología que debe existir entre esos 20-30 nucleótidos que constituyen la secuencia espaciadora y el ADN diana, de manera que la diferencia en un solo nucleótido hará que el sistema no funcione (Ref 4). Por si esto no fuera suficiente, una vez se encuentra homología con la secuencia diana, además debe haber una secuencia de 3-4 nucleotidos en el ADN diana, llamada “Protospacer Adjacent Motif” (PAM), la cual es reconocida específicamente por la nucleasa para unirse y producir el corte en el ADN.
Las potenciales aplicaciones de la técnica son casi ilimitadas pero aquí no hablaremos de la edición genómica que sin duda es una de las más importantes, sino que nos centraremos en la posibilidad de usar CRISPR como nuevos antimicrobianos. Esta aplicación permitirá emplear la tecnología CRISPR para erradicar específicamente patógenos humanos o animales. Esto es, reprogramar CRISPR de manera que la nucleasa ataque específicamente el ADN de un patógeno, (virus o bacteria) cortándolo y creando un daño irreparable que impide que el patógeno sea capaz de sobrevivir. El daño en el ADN es letal, de manera que los mecanismos de reparación de ADN del patógeno no son suficientemente efectivos como para repararlo y por lo tanto muere. Más aún, se puede incluir en el sistema más de una secuencia espaciadora de manera que el sistema CRISPR guiará a la nucleasa a distintos puntos del ADN patógeno produciendo tantos cortes como secuencias espaciadoras se incluyan, aumentando más aún la letalidad y las probabilidades de éxito.
La especificidad de estos sistemas, basada en la perfecta homología de la secuencia espaciadora junto con la secuencia PAM (explicado anteriormente) permitirá actuar con total especificidad sobre el patógeno, sin dañar en absoluto las bacterias comensales que forman parte de nuestro organismo, cosa que no ocurre actualmente con los antibióticos que matan de forma indiscriminada, sin diferenciar entre bacterias “buenas” y bacterias “malas”. Por lo tanto podemos estar seguros que el sistema CRISPR no alterará el resto de poblaciones bacterianas de nuestro organismo ni mucho menos dañará una célula humana. Otra estrategia posible es reprogramar CRISPR para que tenga como objetivo diana el gen de resistencia a antibiótico, que en ocasiones se encuentra codificado en plásmidos y no en el cromosoma del patógeno. De esta manera, una vez destruido el gen de resistencia a antibióticos el patógeno volverá a ser vulnerable a los antibióticos actuales y además se evita la dispersión del gen de resistencia a otras bacterias.
Mientras que en la edición genómica todas las aplicaciones se han basado en la endonucleasa Cas9, parece que las aplicaciones de CRISPR como antimicrobianos están basadas tanto en el uso de Cas9 como en la exonucleasa Cas3. La endonucleasa Cas9 produce un corte en la doble hebra de ADN en un punto único, el cual puede ser reparado y empleado para corregir mutaciones, crear inserciones o delecciones etc. Sin embargo la nucleasa Cas3 a pesar de que sólo corta una de las hebras de ADN tiene actividad exonucleasa, por lo que iniciado el corte continua degradando el ADN, como si de un “comecocos” se tratara, generado por tanto un daño letal irreparable.
Actualmente el uso de CRISPR como nuevos antimicrobianos está siendo estudiado por diversos grupos de investigación en todo el mundo y ha dado lugar a numerosas patentes y a la creación de varias “start-up” de universidades y centros de investigación, orientadas a desarrollar esta tecnología para su aplicación en humanos.
Sin embargo, todavía existen ciertas limitaciones para poder emplear esta tecnología. Las nucleasas son proteínas que actúan dentro de la célula atacando el ADN. Por lo tanto para ser capaces de atacar el ADN de un patógeno debemos ser capaces de hacer llegar el sistema CRISPR dentro del patógeno, esto es, dentro de la bacteria o del virus para que corten su genoma. La actual limitación radica en encontrar mecanismos eficientes para la administración del sistema CRISPR y que este llegue de forma efectiva a los patógenos. Numerosos estudios han demostrado la capacidad de destruir patógenos en condiciones de laboratorio, in vitro, introduciendo el sistema CRISPR dentro de la bacteria patógena mediante procedimientos estándar donde todo ocurre en un tubo de ensayo. Sin embargo esto no se puede aplicar en un paciente por razones obvias. Además, el patógeno puede estar disperso en varios puntos del cuerpo humano o en zonas no accesibles para la aplicación directa del sistema CRISPR. Algunos estudios en animales ya han mostrado la efectividad de CRISPR en infecciones cutáneas y hay quien propone la administración de CRISPR en grandes cantidades mediante una solución o incluso una pomada. Sin embargo, el principal objetivo de la mayoría de los estudios actuales es ser capaces de actuar en el microbioma humano erradicando patógenos intestinales como Clostridium difficile, que causa graves infecciones recurrentes, con una alta tasa de mortalidad, y que se ha convertido en una bacteria multi-resistente a todo tipo de antibióticos. Otros estudios actuales tienen como objetivo diana los patógenos intestinales pertenecientes al grupo Shigella o Salmonella. En todos estos casos, es más difícil aun hacer llegar el sistema CRISPR de manera efectiva al patógeno, ya que tras su administración, debe sobrevivir el paso del estómago, llegar al intestino y una vez allí acceder al interior del patógeno. Las estrategias actuales están basadas en el empleo de fagos, que precisamente son virus capaces de infectar bacterias. De esta manera, podemos usar un virus bacteriano (un fago) como vector del sistema CRISPR para que administrando dicho virus (inocuo para humanos) llegue al intestino, reconozca específicamente la bacteria patógena y le inyecte el sistema CRISPR en su interior.
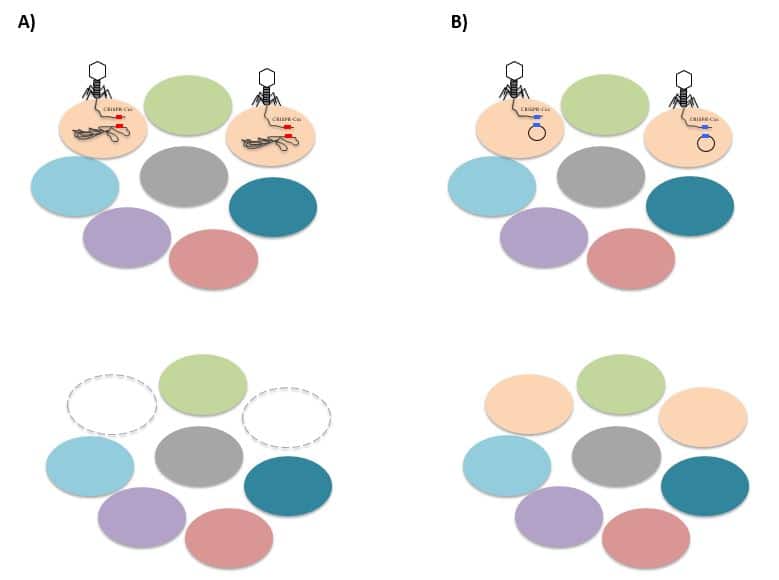
Empleo de fagos para la administración de los sistemas CRISPR-Cas. A) El fago se une específicamente a la bacteria patógena y tras inyectar su material genético (previamente modificado) el sistema CRISPR-Cas es capaz de reconocer el ADN diana, el cromosoma del patógeno y atacarlo produciendo su muerte. B) El fago se une específicamente a la bacteria patógena y tras inyectar su material genético (previamente modificado) el sistema CRISPR-Cas es capaz de reconocer el ADN diana, el gen de resistencia antibiótico codificado en un plásmido, atacarlo y destruirlo, de manera que la bacteria patógena sigue viva pero es sensible al antibiótico.
Toda una obra de ingeniería genética que permitiría erradicar específicamente el patógeno sin alterar el resto de bacterias comensales de la microbiota intestinal. Aunque los resultados preliminares son esperanzadores todavía falta mucho trabajo por desarrollar, inicialmente para llevar a cabo ingeniería genética en fagos capaces de expresar el sistema CRISPR y posteriormente, estudiando las vías de administración para que los fagos lleguen de manera viable a la población diana, el patógeno, permitiendo una eficacia relevante a nivel clínico. El uso de fagos añade todavía mas especificidad al sistema ya que los fagos tienen un rango de hospedador muy concreto, por lo que esto asegura que el fago no infectaría a la bacteria incorrecta, pero a la vez supone una limitación para desarrollar una herramienta efectiva contra diversos patógenos. Actualmente están surgiendo otras propuestas que podrían ser utilizadas como vectores de los sistemas CRISPR; éstas se encuentran en estadios más iniciales de investigación pero que sin duda en los próximos años aportarán grandes resultados.
Como último punto, será interesante ver cómo reaccionan los organismos reguladores ante la aplicación de esta tecnología como nuevo antimicrobiano ya que está basado en la ingeniería genética, y a pesar de la seguridad de su uso es muy posible que levante críticas de los organismos reguladores y, por supuesto, de plataformas anti-GMO. Esperemos que la cordura se imponga y se establezca un marco legal acorde a los avances científicos y al desarrollo de la tecnología, y sobretodo acorde con la necesidad de desarrollar nuevos antimicrobianos que permitan superar las adversidades existentes con los actuales antibióticos.
About the Author: Claudio Hidalgo Cantabrana
5 Comments
Comments are closed.







Hola, rogaria me respondiesen a una duda que tengo. Disculpen mi ignorancia en estos temas. Si el espaciador que hace guia para que la nucleasa corte el adn por el lugar exacto y dicho espaciador es de entre 20 y 30 nucleotidos, al utilizarlo en una celula eucariota, por ejmplo una humana, ¿no es posible que en 3 mil millones de nucleotidos se encuentre esa cadena repetida.?. ¿Cómo sabemos que está cortando en el lugar correcto?. Entiendo que previamente se habrá hecho una busqueda informatica en todo el genoma humano y se sabe de antemano las posbilidades de que haya mas de una coincidencia pero me gustaria que me respondiesen. Gracias
Hola «javierxrq», soy Claudio Hidalgo el autor del artículo. Efectivamente tienes razón en lo que dices pero vamos por partes. El primer punto de especificidad del sistema CRISPR es el PAM (Protospacer Adjacent Motif), que es una secuencia de 3 nucleótidos que es reconocida por la polimerasa y permite la unión de la polimerasa al ADN. Tras este reconocimiento y unión, si hay homología en los 20 nucleotidos adyacentes con los 20nt que se han usado como guía entonces la nucleasa cortará el ADN, sino no.
Para diseñar una diana, lo primero que hay que hacer es localizar en el gen de interés a modificar esa secuencia PAM de 3 nucleótidos, que es «NGG» si se usa la nucleasa típica Cas9 de S.pyogenes. Localizada este NGG, los 20 nucleotides adyacentes serán los que se sinteticen químicamente y se usen como guía de la nuclesasa para ir a cortar en ese punto. Efectivamente hay que hacer un análisis bioinformático previo y ver la posibilidad de que esa secuencia de 20+3 no esté presente en otra región del genoma para estar muy seguros que no se va a modificar una región no deseada.
Espero haberte ayudado.
Vale. Gracias por tu respuesta pero permiteme seguir. Y si el gen que buscamos no codifica ninguna aminoacido NGG? Cómo lo localizamos entonces? Cambiariamos de PAM? Habria que usar otro Cas distinto? O quizas todos los genes codifican al menos un AGG, TGG,GGG, O CGG y entonces no hay problema, pero creo que no todos tendran ese PAM esencial para localizar el sitio de corte.
Por otro lado ya que estoy, los que nos interesa la biologia molecular pero no hemos estudiado esto, sino derecho, nos gustaria un post explicando, como se hace, por ejemplo la busqueda, corte y edicion de un gen en un laboratorio. A los que no entendemos nos parece un misterio y no sabemos si veis los genes en algun microscopio, que ya sé que no, o utilizais un secuenciador o solo tubos de ensayo. Y tambien como comprobais luego los resultados de dicha edicion genomica.
Muchas gracias. Espero vuestras respuestas.
Hola Javierxrq, disculpa pero no había visto este comentario. La probabilidad de que exista un motivo «NGG» en cualquier gen es bastante alta por lo tanto no es una limitación para aplicar la tecnología. Esta secuencia PAM depende de la nucleasa Cas9 utilizada. El PAM «NGG» es reconocido por la nuclease Cas9 de Streptococcus pyogenes, (SpyCas9) que fue la primera en ser caracterizada, usada y aplicada worlwide desde bacterias a humanos, precisamente por ser la primera que se caracterizó pero no es la única ni posiblemente la mejor. Si el gen a modificar no tiene el dominio NGG entonces tendremos que buscar otra nuclease Cas9 de otra bacteria distinta, muchas bacterias la tienen, eso si tendremos que caracterizar el PAM de esta Cas9 y los otros elementos necesarios para que funcionen, el guideRNA principalmente.
[…] realizar esta edición de genes, He Jiankui utilizó la tecnología CRISPR-CAS9 de la que os hemos hablado en multitud de ocasiones. Se trata de, en primer lugar, proceder a una fecundación in vitro y […]